封面|Science封面重磅:新型AI算法准确预测RNA三维结构( 二 )
解析 RNA 三维结构序列决定结构,结构决定功能,这是 RNA 和蛋白质这样的生物大分子的基本法则。这也是为什么科学家热衷于研究这些生物分子机器精细结构的深层原因,因为只有知道了结构才能知道功能背后的原理。
长期以来,科学家对 RNA 复杂多样的结构并不十分了解。DNA 是能预测的双螺旋结构,RNA 与 DNA 不同,是单链折叠成的隆起、假结、头样、发夹等多种多样的复杂三维循环结构。满足不同功能状态的需要,不同折叠能相互转化。科学家对 RNA 的上述信息了解非常肤浅,这也是目前 RNA 功能研究中最薄弱的环节。
近年来,科学家开始对 RNA 结构研究发起挑战。Bevilacqua, Weissman 等设计的技术能对细胞内大量 RNA 结构进行整体解析,初步研究结果发现,活细胞内 RNA 折叠方式与人工条件下的完全不同。
文章插图
图 | RNA 3D 结构测定与分析(来源:NIH)
RNA 分子通常有一个线性核苷酸链,但在细胞核内合成后,会通过自身核苷酸配对迅速折叠,然后进一步折叠成复杂三维结构,与蛋白和其他 RNA 分子发生相互作用时会改变形状。
研究 RNA 结构的大部分技术利用核苷酸相互结合的特点,或者序列对某些酶的敏感性。计算机模拟技术也有助于整体结构的分析。但是这些方法非常繁琐,一次只能分析一个分子的一部分。
因此,虽然越来越多的证据表明,从细菌到人类功能性 RNA 无处不在,理论上靶向 RNA 可以治疗人类任何疾病,但是由于无法准确获得 RNA 的三维结构,有关 RNA 的基础研究和药物拓展一直受阻。
AI 算法解决结构生物学挑战预测蛋白质复合物以及 RNA 三维空间结构是生物化学领域一项巨大的挑战,对于基础科学和药物研发具有重大的意义。Stephan Eismann 博士等人致力于相关的研究。
以往蛋白质复合物以及 RNA 等生物大分子结构人工智能预测模型通常会直接利用给定的结构特征来进行训练。然而,在研究过程中,人们发现,这种利用上述方法开发出的模型预测结果往往偏向于模型训练时喂养结构的特征。因此,即使科学家努力试图对模型进行优化,但是预测的准确性依旧十分有限。
为了准确预测蛋白质复合物以及 RNA 等生物大分子的三维结构,Eismann 博士等人换了一个角度思考,即能否直接利用人工智能学习上述生物大分子原子水平的结构,以提高预测结果的准确性。
为此,研究人员基于深度学习网络开发了一种全新的人工智能模型,ARES。与其他模型不同,ARES 旨在准确了解生物大分子中每个原子的准确位置和几何排列,以及不同原子之间的相对位置。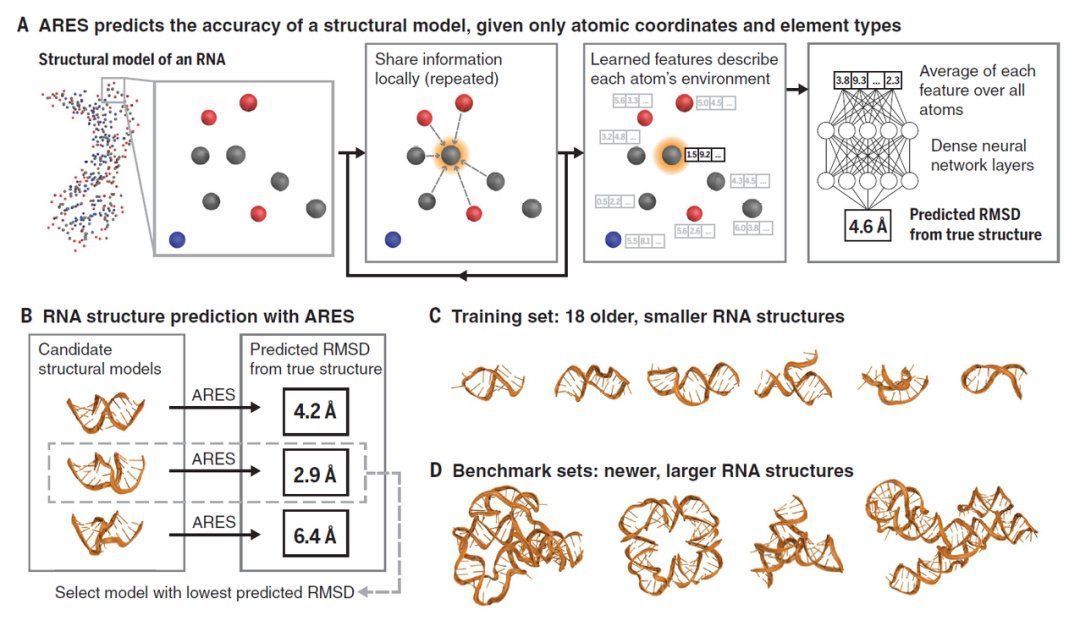
文章插图
图 | ARES 模型(来源:Science)
早在 2020 年的时候,Eismann 博士等人就曾利用这一理论成功开发出了一种准确预测蛋白质复合物三维结构的模型,相关研究发表在 Proteins 杂志上。
而在本研究中,Eismann 博士等人继续对 ARES 进行了优化。研究人员采用原子坐标作为输入数据,而不包含 RNA 结构的空间特征。通过不断调整参数,ARES 首先了解每个原子的功能和空间排列,随后识别碱基配对规则、RNA 螺旋最佳几何形状以及三维空间结构。
文章插图
图 | ARES 可以准确识别出正确的 RNA 分子 3D 形状(来源:斯坦福大学官网)
最终,研究人员仅用了 18 个已知 RNA 三维结构,就成功训练出了 ARES。在随后的研究中,Eismann 博士等人证实,ARES 虽然仅由 18 个 RNA 结构训练而来,但是它同样可以准确预测其他复杂 RNA 的三维空间结构,且准确性均显著优于既往的模型。
- text|《2021大数据产业年度创新技术突破》榜重磅发布丨金猿奖
- Java|重磅丨屯粮积草网与腾讯达成2022年度战略合作,实现主流搜索引擎全覆盖!
- 创新大赛|RTE创新大赛三强揭晓 !《实时互动场景创新生态报告》重磅发布
- 生意|兼职月入10万:红包封面的暴利生意
- 地球上的我们正在经历一个前所未有的历史演变也许|预告|智慧地球之元宇宙重磅来袭,1月15日下午14时全网直播
- 姚晨简约质感开年封面出炉 时尚大气魅力满分
- 新年一大重磅喜讯,天问一号从火星传回信号,画面公开后震惊全球
- 微信红包|微信发布 8.0.18 正式版,朋友圈、微信红包封面都变了
- 蓉贝|第十九届中国国际软件合作软洽会在蓉举行 多个重磅项目落地成都高新区
- var|微信发布 8.0.18 正式版,朋友圈、微信红包封面都变了
